Зачем биомедикам понадобился свой язык описания онтологий, как эти задачи решает BioCypher и при чем здесь Neo4j: практическое приложение Data Science и графовых алгоритмов в биомедицинской сфере.
Что такое BioCypher
Графовые алгоритмы активно применяются в биомедицине для анализа различных биологических данных, таких как геномные, протеомные, данные о белковых взаимодействиях и пр. Однако, построение графа знаний для биомедицинских задач обычно занимает очень много времени. Ускорить процесс создания графа биомедицинских знаний поможет BioCypher – мощная платформа обработки, анализа и прогнозирования биомедицинских данных, разработанная компанией Cypher Genomics. BioCypher объединяет в себе методы машинного обучения, биоинформатику и генетику для анализа геномных данных, чтобы предоставить своим пользователям инструменты для обработки и интерпретации этих данных. В основе BioCypher лежит модульая концепция: модульность источников данных, модульность структурирующей онтологии и модульность выходных форматов. Такая конструкция обеспечивает высокую степень гибкости и возможности повторного использования.
BioCypher включает три основные области построения графа знаний для биомедицины:
- Улучшенное взаимодействие с ресурсами за счет передачи любых данных в инфраструктуру с помощью ET-подобных процессов. Различные адаптеры помогают настроить выбранные данные, позволяя пользователю самому определить нужные данные на уровне узла или ребра. Пользователь загружает нужные данные, а конвейер сам помещает их в место назначения. Адаптер BioCypher представляет собой Python-программу, которая отвечает за подключение к ядру BioCypher и предоставляет ему данных из связанного с ним ресурса. Адаптер должен соответствовать интерфейсу, определенному ядром, чтобы предоставлять информацию об узлах и ребрах, для автоматической гармонизации содержимого.
- Онтология — формальное представление области знаний, иерархическая структура понятий и отношений. Понятия организованы в иерархию, где каждое понятие является подклассом более общего понятия. Такая подготовленная экспертами информация сопоставляется с данными, а пользователь может выбрать нужные ему онтологии для исследований. BioCypher сопоставляет любые входные данные с базовой онтологией высокоуровневой модели Biolink. Эта модель данных биологических объектов (гены, болезни, фенотипы, пути, особи, вещества и пр.) и их ассоциации разработана как способ стандартизации типов и реляционных структур в графах знаний, которые могут быть графом свойств или RDF-хранилищем триплетов. Схема данных в модели Biolink выражается в виде YAML-документа и предполагает граф свойств, где узлы представляют отдельные объекты, а ребра представляют отношения между ними. Модель Biolink предоставляет схему для представления как узлов, так и ребер. Например, концептуальный белок представлен белком класса Biolink . Чтобы ввести белки в граф знаний, нужно просто определить компонент узла с помощью метки класса белок.
- Вывод – объединенные данные экспортируются в Property Graph, SQL или RDF, чтобы пользователи могли с ними работать, в т.ч. совместно и повторно использовать схему данных. Изначально разработка BioCypher была сосредоточена вокруг выходных данных графовой базы данных Neo4j из-за миграции база данных о белковых взаимодействиях OmniPath на серверную часть Neo4j. Однако, разработчики представляют BioCypher как абстракцию процесса построения графа биомедицинских знаний, а потому стремятся подключить другие форматы вывода, такие как RDF, SQL и ArangoDB.
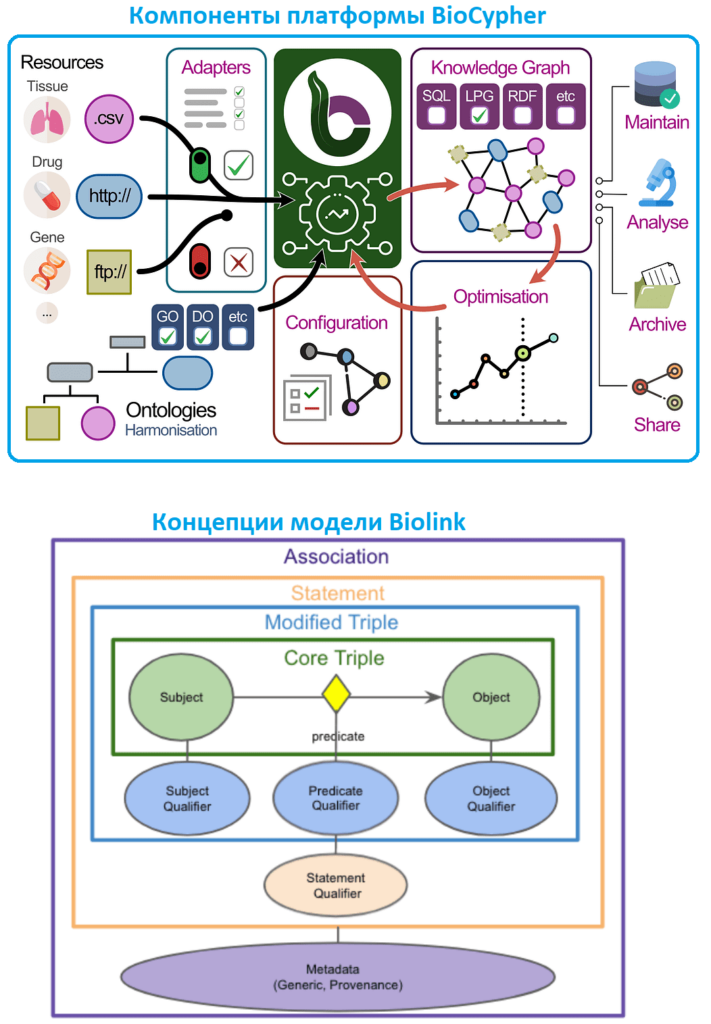
Познакомившись с основными концепциями BioCypher, далее рассмотрим, как эта платформа технически связана с нативной графовой базой данных Neo4j.
Взаимодействие с Neo4j
Драйвер BioCypher является основным подмодулем BioCypher. Он устанавливает соединение с работающей базой данных графов через neo4j.GraphDatabase.driver, интегрирует функции других подмодулей и служит внешним интерфейсом BioCypher. Driver – это основной класс для взаимодействия с BioCypher в классе адаптера хост-модуля. Он выполняет аутентификацию и базовое управление базой данных, а также создание и манипулирование записями графа. Его можно создать и получить к нему доступ следующим образом:
import biocypher d = biocypher.Driver( db_name = "neo4j", db_user = "neo4j", db_passwd = "neo4j" )
Драйвер создается при инициализации адаптера, а затем вызывается для взаимодействия с работающим графом и для экспорта полной базы данных в формате CSV для функции административного импорта Neo4j . При создании экземпляра он автоматически оценивает базу данных графов, к которой он подключен, что указывается с помощью аргумента db_name. Если база данных уже содержит граф BioCypher, определяется структура этого графа. Если в текущей активной базе данных нет графа BioCypher или если пользователь явно указывает это с помощью wipe-атрибута драйвера, создается новая база данных BioCypher с использованием конфигурации схемы, указанной в YAML-файле конфигурации schema-config.yaml.
Для быстрого перехода на BioCypher отдельные термины или целые запросы могут быть переведены с использованием информации, представленной в файле schema_config.yaml. Доступ к функциям перевода можно получить с помощью методов драйвера biocypher.driver.translate_term(), biocypher.driver.reverse_translate_term(), biocypher.driver.translate_query() и biocypher.driver.reverse_translate_query(). Например, чтобы узнать обозначение отношений «ген_ген» в BioCypher, можно вызвать команду:
driver.translate_term("gene_gene")
Узнать исходное название отношений PostTranslationalInteraction поможет вызов обратной функции:
driver.reverse_translate_term("PostTranslationalInteraction")
После запуска драйвер BioCypher можно использовать для изменения текущего графа, добавляя или удаляя узлы, ребра, свойства, ограничения и пр., используя методы biocypher.driver.Driver.add_nodes() и biocypher.driver.Driver.add_edges()для внесения новых записей в графовую базу данных.
После записи файлов графа знаний с помощью BioCypher пригодится CLI-инструмент neo4j-admin как вызов командной строки, необходимый для импорта данных в Neo4j:
bc.write_import_call()
Это создает исполняемый сценарий оболочки в выходном каталоге, который можно выполнить из папки базы данных или скопировать в терминал Neo4j для импорта графика в графовую БД. Поскольку BioCypher создает отдельные файлы заголовков и данных для каждого типа объекта, вызов импорта удобно объединяет эту информацию в одну команду, детализируя расположение всех файлов на диске, поэтому нет необходимости копировать данные.
Как мы уже отмечали в прошлой статье, Neo4j может управлять несколькими проектами, каждый из которых имеет несколько экземпляров СУБД. В свою очередь, каждый экземпляр СУБД может содержать несколько баз данных, с которыми можно оперировать вместе благодаря концепции составных БД. Это особенно удобно при работе с огромными объемами биомедицинских данных в различных онтологиях, поскольку кроме Biolink, существует еще множество специализированных онтологий, например, Sequence Ontology для детального представления вариантов последовательностей, и MONDO для детального представления болезней.
Будучи написанной на Python, который вполне можно назвать главным языком в Data Science, платформа BioCypher является расширяемой и адаптируемой. В частности, можно изменить базовую модель используемой онтологии, добавив собственный класс, метод или интерфейс.
В заключение отметим несколько наиболее крупных биомедицинских проектов, реализуемых с помощью BioCypher:
- Проект «Влияние геномной изменчивости на функцию» (IGVF) строит обширный граф биологических знаний, чтобы попытаться связать человеческие вариации и болезни с наборами геномных данных на уровне отдельных клеток. Исследователи создают ориентированный на пользователя API и GUI, который будет обращаться к этому графу. BioCypher выступает посредником между Biolink и графовой СУБД ArangoDB, предоставляя структуру, которую можно использовать для анализа десятков файлов данных и форматов в схему Biolink.
- CROssBAR (Comprehensive Resource of Biomedical Relations) — комплексный ресурс биомедицинских взаимосвязей с приложениями глубокого обучения и представлениями графов знаний. Это комплексная система, которая объединяет крупномасштабные биомедицинские данные из различных ресурсов и сохраняет их в NoSQL-СУБД, обогащает эти данные прогнозированием отношений между многочисленными биомедицинскими объектами на основе глубокого обучения, тщательно анализирует обогащенные данные для получения биологически значимых модулей и отображает их пользователю с помощью легко интерпретируемых, интерактивных и разнородных графов знаний в рамках открытого доступа онлайн-сервиса.
- Проект построения графа знаний, который включает в себя атрибуты метаболитов, белков и их взаимодействий для изучения межклеточных коммуникаций. Для построения графа знаний используются данные из нескольких баз, которые предоставляют информацию в различных форматах. BioCypher принимает все эти входные данные, придает им воспроизводимую структуру и обеспечивает правильное управление версиями. Граф знаний, созданный BioCypher, можно связать с биологическими вопросами, касающимися конкретных тканей, заболеваний или свойств метаболитов, что облегчает последующий анализ и интерпретируемость.
Узнайте больше про использование графовых алгоритмов и средств работы с ними для практического применения в реальных проектах аналитики больших данных на специализированных курсах нашего лицензированного учебного центра обучения и повышения квалификации для разработчиков, менеджеров, архитекторов, инженеров, администраторов, Data Scientist’ов и аналитиков Big Data в Москве:
Источники

 635
635 

